Formation metabarcoding 2019
Contexte
Le séquençage à haut débit des amplicons de marqueurs taxonomiques tels l’ARN ribosomique, les gènes COI/COX ou les ITS a ouvert de nouveaux horizons dans l'étude des communautés de macro et micro-organismes.
Le but de cette formation est, d’une part, d’introduire les concepts clés liés aux analyses de metabarcoding et les illustrer au moyen de cas concrets d’analyse et, d’autre part, de former les utilisateurs aux traitements de données de metabarcoding au travers de l’usage du logiciel FROGS ainsi que d’autres outils d’analyses (DADA2, R scripts).
Objectifs
Le principal objectif est d’initier les participants à la pratique de l’outil FROGS utilisé pour analyser les données de séquençage à haut débit de metabarcoding. Le cours sera basé sur des exercices théoriques et pratiques.
Le second objectif de ces journées sera d’apporter un complément d’information sur des cas concrets d’analyse de diversité au moyen d’exposés d’experts du domaine.
Le troisième est de proposer des moments d'échanges conviviaux entre les participants et les intervenants autour des repas du midi et du soir inclus dans cette formation.
Dates et lieu
- Du 2 au 6 décembre 2019
- Station Biologique de Roscoff
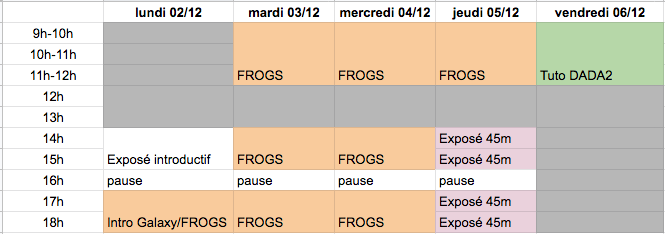
Programme
- La formation FROGS sera assurée par :
- Olivier Rué (INRA, Jouy en Josas)
- Cédric Midoux (IRSTEA, Antony)
- Mahendra Mariadassou (INRA, Jouy en Josas)
- Différents tutoriaux et exposés autour du metabarcoding seront assurés par :
- Olivier Chapleur (IRSTEA, Antony) :
"Les analyses de diversité microbienne au service d’une meilleure compréhension des écosystèmes microbiens complexes" - Raffaele Siano (IFREMER, Brest)
- Marjorie Couton (Station Biologique, Roscoff) :
"Le metabarcoding appliqué à la détection et au suivi des espèces non-indigènes marines" - Angélique Gobet (IFREMER, Palavas) :
"L'écologie microbienne en aquaculture" - Charles Bachy (Station Biologique, Roscoff) :
"Les prémices du metabarcoding par séquençage nanopore"
- Olivier Chapleur (IRSTEA, Antony) :
Public visé
Doctorants, ITA, chercheurs, enseignants et ingénieurs impliqués dans des projets concrets d’analyse de données de metabarcoding.
Pré-requis
Avoir une connaissance de l'environnement Galaxy et un projet d'analyse de données de metabarcoding.
Venir avec son ordinateur portable.
Nombre de participants attendus
23 participants.
Etant donné le nombre limité de places pour cette formation, une sélection des participants sera réalisée dans le cas où nous aurions reçu plus de 23 candidatures.
Frais d'inscription
600€ HT (tarif unique)
Ces frais d'inscription comprennent les déjeuners et diners qui seront pris au restaurant Gulf Stream à Roscoff.
Organisateurs
- Laure Quintric (Service de Bioinformatique, IFREMER, Brest)
- Patrick Durand (Service de Bioinformatique, IFREMER, Brest)
- Erwan Corre (Plateforme ABiMS, CNRS-Sorbonne Université, Roscoff)
Inscription
Les inscriptions sont désormais closes.